DDI Induction
L'induction du cytochrome P450 fait partie des services ADME expérimentaux in vitro. Le profil d’induction d’un nouveau composé à tester doit être systématiquement évalué.
Ces études incluent, mais ne sont pas limitées à:
- CYP1A2, CYP2B6 et CYP3A4
- Autres enzymes de phase I, notamment la monoamine oxydase (MAO), la flavine monooxygénase (FMO), la xanthine oxydase (XO) et l’alcool / aldéhyde déshydrogénase
- Enzymes de phase II, y compris les UDP glucuronosyl transférases (UGT)
Les hépatocytes humains constituent le système d’essai principal pour évaluer l'induction enzymatique in vitro, à la fois avec des hépatocytes humains fraîchement isolés et des hépatocytes cryoconservés disponibles pour un usage courant.
Lors de la détermination du potentiel d'induction enzymatique de nouveaux composés à tester à l'aide d'hépatocytes humains ou de cellules HepaRG™ en culture, il convient de prendre en compte les éléments suivants:
- Variabilité interindividuelle
- Modification du niveau d'ARNm des gènes cibles en tant que critère d'évaluation (le niveau d'activité est également évalué à Eurosafe)
- L'inclusion dans l'expérimentation de contrôles de véhicule, de contrôles positifs (généralement des inducteurs connus) et de contrôles négatifs (généralement des non-inducteurs connus).
Initialement, l’induction du CYP1A2, du CYP2B6 et du CYP3A est évaluée (recommandations de la FDA concernant les études de DDI).
Si aucune induction des enzymes CYP3A4 / 5 n'est observée, il est inutile d'évaluer le potentiel d'induction des enzymes CYP2C, car les enzymes CYP3A4 / 5 et CYP2C sont induites par l'activation du récepteur nucléaire pregnane X (PXR).
Si une induction du CYP3A4 / 5 se produit, toutefois, il est alors nécessaire d'évaluer le potentiel d'induction du CYP2C.
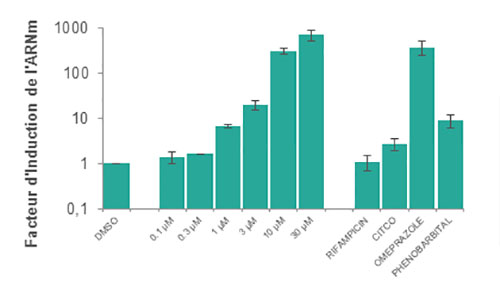
Nous examinons la variation des taux d'ARNm de l'enzyme CYP lors de l'incubation avec le composé à tester en utilisant les seuils décrits dans les directives de la FDA.
Eurosafe évalue également la liaison du médicament aux protéines microsomales et aux cellules afin d’évaluer la fraction libre (fumic, fuhep) et de prédire le potentiel d’interaction hépatique ou d’interaction médicamenteuse in vivo, en utilisant des données métaboliques microsomales in vitro.
DDI Inhibition
L'inhibition des enzymes du métabolisme (CYP et UGT) par un nouveau composé à l'essai peut réduire le métabolisme des médicaments co-médicamentés. Le potentiel de ce composé à tester pour inhiber les enzymes est habituellement étudié pour connaitre les mécanismes d'inhibition précis (réversible ou dépendant du temps) ainsi que le pouvoir d'inhibition (par exemple, Ki). Lien vers FDA guidelines.
Les microsomes hépatiques sont recommandés pour les dépistages et les évaluations mécanistiques. En guise d'alternative aux approches systématiques, des hépatocytes humains ou des cellules HepaRG™ peuvent être utilisés pour déterminer le potentiel d'inhibition du CYP des composés à tester dans l'environnement cellulaire.
Les tests de screening sont effectués avec jusqu'à 9 enzymes du cytochrome P450 (1A2, 2A6, 2B6, 2C8, 2C9, 2C19, 2D6, 2E1, 3A4 / 5) en utilisant 10 réactions de sonde. Le test d'inhibition directe fournit des valeurs de IC50 pour chaque enzyme du CYP. Le test d'inhibition en fonction du temps fournit des valeurs de IC50 directes et décalées pour chaque enzyme du CYP.
Après la détermination de la IC50, nous déterminons le Ki pour le composé à tester par rapport à l'enzyme appropriée. Ce paramètre indique le potentiel d’inhibition ainsi que le type d’inhibition. Ces informations peuvent être utilisées pour estimer l'impact de toute interaction potentielle in vivo.
Eurosafe évalue également la liaison du médicament aux protéines microsomales et aux cellules afin d’évaluer la fraction libre (fumic, fuhep) et de prédire le potentiel d’interaction hépatique ou d’interaction médicamenteuse in vivo, en utilisant des données métaboliques microsomales in vitro.
DDI Phenotyping
Afin de prédire les interactions médicamenteuses, il est essentiel de comprendre quelles enzymes (phase I et phase II) sont responsables du métabolisme d’un composé d’essai donné.
La FDA reconnaît trois méthodes bien caractérisées pour identifier les différentes enzymes du CYP impliquées dans le métabolisme d’un médicament.
- Méthode 1 utilisation d’une banque de microsomes hépatiques humains caractérisée par une activité CYP
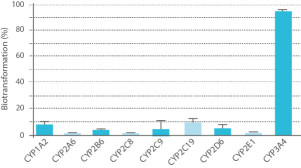
- Méthode 2 utilisation des enzymes CYP recombinantes humaines individuelles
- méthode 3 utilisation de produits chimiques ou anticorps spécifiques comme inhibiteurs d’enzymes spécifiques
- Méthode 3 bis: Eurosafe propose l'utilisation de Silensomes™.
Les Silensomes™ sont des pools validés de microsomes hépatiques humains, qui sont inactivés chimiquement et irréversiblement pour un CYP450 spécifique à l’aide d’inhibiteurs basés sur le mécanisme (MBI). Cette approche permet un phénotypage quantitatif en déterminant la contribution métabolique (fm) au lieu de l’évaluation RAF.
- Yannick Parmentier, Corinne Pothier, Audrey Delmas, Fabrice Caradec,Marie-Michèle Trancart, Fabrice Guillet, Belkacem Bouaita, Christophe Chesne, J. Brian Houston & Bernard Walther, "Direct and quantitative evaluation of the humanCYP3A4 contribution (fm) to drug clearance usingthe in vitro SILENSOMES model", Xenobiotica, Volume 47, pages 562-575, 2016.
- Yannick Parmentier, Corinne Pothier, Nicola Hewitt, Ludwig Vincent, FabriceCaradec, Jia Liu, Feifei Lin, Marie-Michèle Trancart, Fabrice Guillet, BelkacemBouaita, Christophe Chesne & Bernard Walther, "Direct and quantitative evaluation of the majorhuman CYP contribution (fmCYP) to drugclearance using the in vitro Silensomes™ model", Xenobiotica, Volume 49, pages 22-35, 2018.


